² 用于确定植物基因转化条件的表达载体pKAFCR21
特点:pKAFCR21是在农杆菌双核载体pIG121-Hm的基础上,插入pBIsGFP的sGFP绿色荧光蛋白报告基因,以及精简的35S启动子、Ω转录增强子和NOS终止子改造而成的,具有两个独立表达的卡那霉素NPT II耐性基因和潮霉素HPT耐性基因,以及两个独立表达的GUS葡萄糖苷酸酶和sGFP绿色荧光蛋白报告基因。同时含有两个抗生素耐性基因,可以探讨转基因过程中使用抗生素的种类、浓度,选拔时间等条件。同时含有两个报告基因,可以利用化学检测(蓝色)和光学检测(荧光)转基因过程,提高基因转化和选拔效率。
用途:可用于确定植物基因遗传转化条件,如探讨农杆菌菌株,共培养条件,抗生素种类、浓度,选拔时间等对基因转化效率的影响。
制作人:陈任
使用方法:参考以下论文
Ren Chen*, Mayumi Gyokusen, Yoshihisha Nakazawa, Yinquan Su, Koichiro Gyokusen. Establishment of an Agrbacterium-mediated transformation system for Periploca sepium Bunge. Plant Biotechnol. 2010, 27: 173-181

² 用于植物单基因表达载体构建的质粒pNULPGE200
特点:为克服目前常用于植物基因表达载体构建的质粒由于酶切位点有限,目的基因片段难于插入和连接,缺少植物基因表达所必须的启动子、终止子、筛选标记等功能元件的缺点,构建了一个用于植物单基因表达载体构建的质粒载体pNULPGE200。该质粒载体引入了植物基因表达最常用的CaMV 35S启动子和NOS终止子,以及之间的多克隆酶切位点MCS。利用pNULPGE200构建植物基因表达载体,经PCR等方法克隆得到的目的基因可以直接连接到35S启动子与NOS终止子之间,使目的基因能够在植物体内稳定表达;同时该质粒载体还具有独立表达的卡那霉素NPT II耐性基因和sGFP绿色荧光蛋白报告基因,可用于基因转化时的筛选。
用途:植物单基因表达载体构建用质粒,可用于植物单基因遗传转化、基因功能鉴定等。
制作人:陈任
使用方法:参考以下论文和专利。
安韶雅, 虎娟, 张虹, 孙放, 马霞, 王晨, 陈任*. 用于植物基因表达载体构建的质粒改造及其应用. 生命科学研究, 2018, 22(2): 114-121
用于植物基因表达载体构建的质粒载体及其应用(CN201710360919.5),安韶雅、虎娟、周涛、林哲、张虹、陈任,中国国家知识产权局,2017.05.26

² 用于植物多基因表达载体构建的质粒系统pKAFCR80和pKAFCR100
特点:为克服目前常用于植物基因表达载体构建的质粒由于酶切位点有限,目的基因片段难于插入,缺少植物基因表达所必须的启动子、终止子和筛选标记等功能元件,无法构建多个基因表达载体等缺点,通过对大肠杆菌质粒pUC18和双核农杆菌Ti质粒pBI121进行改造,建立了一套适用于植物基因表达载体构建的质粒系统,即重组质粒pKAFCR80和pKAFCR100。pKAFCR80具有植物基因表达最常用的CAMV 35S启动子和NOS终止子,并在其上游、中间和下游引入了多个克隆酶切位点,利用PCR等方法克隆得到的目的基因片段可以多种方式连接到35S启动子与NOS终止子之间;pKAFCR100具有卡那霉素NPT II耐性基因和sGFP绿色荧光蛋白报告基因,以及其中间的多克隆酶切位点。pKAFCR80与pKAFCR100两个质粒的配合使用,可以方便地构建植物单个或多个基因表达载体。
用途:植物多基因表达载体构建用质粒,可用于植物多基因遗传转化、基因功能鉴定等。
制作人:陈任
使用方法:参考以下论文和专利。
陈任*, 张虹, 张芮, 虎娟, 周涛, 安韶雅, 林哲. 用于植物多基因表达载体构建的质粒系统. 分子植物育种, 2018, 16(4): 1138-1146
用于植物多基因表达载体构建的质粒体系及其应用 (CN201610767834.4),陈任、张虹、张芮、周涛、安韶雅,中国国家知识产权局,2016.12.21

² 用于植物基因融合蛋白载体构建的质粒pNULPGE300
特点:pNULPGE300是在农杆菌双核载体pBI121的基础上,插入sGFP绿色荧光蛋白报告基因改造而成的。目的基因(去除终止子)只需连接到Xba I和Sma I(Xma I)酶切位点之间,可与下游的sGFP绿色荧光蛋白报告基因形成融合表达结构,基因转化后通过荧光(荧光显微镜或者共聚焦显微镜)可以观察到基因编码的蛋白质在植物体内的亚细胞定位。该质粒载体具有独立表达的卡那霉素NPT II耐性基因,可用于基因转化时的筛选。
用途:可用于确定基因编码的蛋白质在植物体内的亚细胞定位、共聚焦显微镜观察。
制作人:陈任

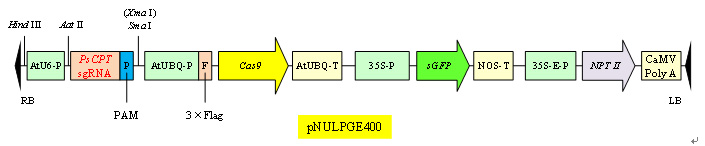
² 用于植物基因编辑(CRISPR/Cas9)载体构建的质粒pNULPGE400
特点:pNULPGE400是在农杆菌双核载体pCAMBIA2300-Cas9的基础上,插入35S启动子、sGFP绿色荧光蛋白报告基因、NOS终止子,并引入多个酶切位点改造而成的。该质粒载体具有独立表达的卡那霉素NPT II耐性基因和sGFP绿色荧光蛋白报告基因,前者可用于基因转化时的抗生素筛选,后者可以利用光学检测(荧光)转基因过程,提高基因转化和选拔效率。目的基因的sgRNA靶点序列(包括PAM序列),只需连接到Aat II和Sma I(Xma I)酶切位点之间,可与下游的Cas9切口酶形成CRISPR/Cas9基因编辑结构,通过基因转化可以敲除该目的基因(使两个相近的切口造成DNA双链断裂)。
用途:植物基因编辑载体构建用质粒,可用于植物基因敲除、基因功能鉴定等。
制作人:陈任
使用方法:
1. 根据目的基因的外显子序列(CDS部分)进行sgRNA靶点设计:使用网站http://crispr.mit.edu或http://crispr.hzau.edu.cn/cgi-bin/CRISPR2/SCORE进行预测,并分析预测靶点的脱靶位点数,避免基因脱靶产生。
2. 分析sgRNA靶点序列:使用网站 http://www.oligoevaluator.com,综合分析sgRNA 靶点的 Tm值(56-62°C)、GC含量(45-60%)及二级结构,最终确定适宜的 sgRNA 序列。
3. 合成sgRNA序列,连接到载体:合成2条互补的sgRNA序列,加上接头或酶切位点,退火形成双链DNA,然后连接到载体。
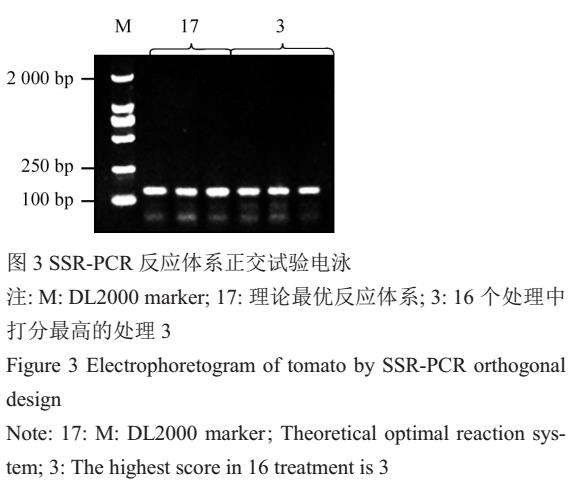
² 番茄叶片DNA提取、SSR-PCR反应体系优化及引物筛选
特点:利用高通量组织研磨机研磨微量叶片,通过改良CTAB法快速提取DNA,并利用L16(45)正交设计试验,综合采用直观量化分析和方差分析两种方法,分析Mg2+、dNTPs、Taq DNA聚合酶、引物、模板DNA浓度5个因素对番茄SSR-PCR扩增的影响,比较不同处理组合扩增效果的差异,最终筛选与验证最优的番茄SSR-PCR反应体系,并在此体系下对SSR引物进行筛选。结果表明番茄SSR-PCR最佳反应体系(20 µL)为Mg2+ 3.0 mmol/L、dNTPs 0.4 mmol/L、Taq DNA聚合酶0.5U、引物0.5 µmol/L、模板DNA 40 ng;且利用优化后的反应体系,从540对SSR引物中筛选出373对(占69.07%)扩增条带清晰、多态性丰富的引物。该SSR-PCR体系的建立为番茄种质资源遗传多样性分析,品种鉴定及指纹图谱构建等研究提供了一个标准化的程序。
制作人:王晓敏、李建设
使用方法:参考以下论文
芮文婧,王晓敏*,张倩男,张学琴,吕原,胡学义,高艳明,李建设*. 番茄叶片DNA提取、SSR-PCR反应体系优化及引物筛选. 分子植物育种, 2018, 16(7): 2230-2236
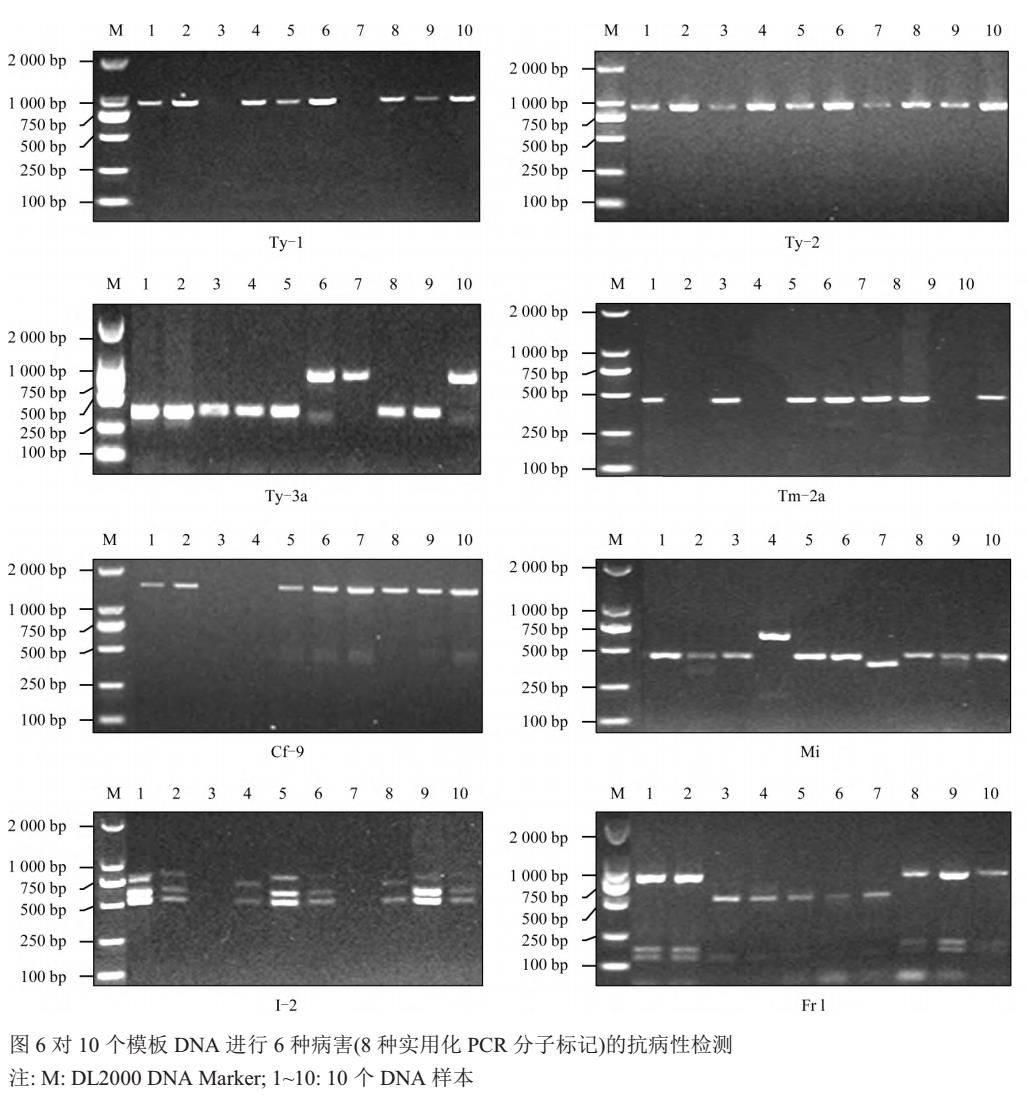
² 番茄叶片DNA提取、SSR-PCR反应体系优化及引物筛选
特点:利用抗根结线虫病Mi标记,在L16 (45)正交设计试验基础上,采用直观量化分析和方差分析两种方法,对番茄实用化PCR分子标记PCR反应的5个因素(引物,Mg2+,dNTPs,模板DNA和Taq酶)进行优化;随后分别筛选黄化曲叶病毒病、烟草花叶病毒病、根结线虫病、枯萎病、叶霉病和颈腐根腐病6种病害的8个实用化PCR分子标记PCR程序的退火温度;并对循环次数进行筛选;最后利用优化后的PCR体系与程序对供试的1442份番茄材料(包括番茄种质资源,群体材料,组合及品种)进行抗病性鉴定。结果表明番茄实用化PCR分子标记的PCR最佳反应体系(20 µL)为引物0.5 µmol/L、Mg2+ 1.5 mmol/L、dNTPs 0.3 mmol/L、DNA 480 ng DNA、聚合酶1.0 U Taq;PCR程序中8个分子标记共同最适退火温度为56℃,最佳循环次数为33次;筛选出585份多抗(含2种以上的抗病基因)番茄材料,可作为中间材料或亲本材料进行多抗品种选育。该实用化PCR分子标记的PCR体系的建立为番茄利用抗病基因分子标记检测与筛选多抗种质资源、品种或群体材料提供了标准化的程序。
制作人:王晓敏、李建设
使用方法:参考以下论文
王晓敏,张倩男,芮文婧,吕原,高艳明,李建设*,张亚红. 番茄实用化PCR分子标记反应体系与程序优化. 分子植物育种, 2018, 17(4): 1238-1248